随着生物学研究的不断深入,单细胞分析已经成为揭示细胞异质性、理解疾病机制和开发精准治疗的重要手段。从癌症到神经退行性疾病,单细胞RNA测序(scRNA-seq)和单细胞基因组学等技术的快速发展为我们带来了前所未有的研究视角。随着单细胞技术的飞速发展,数据量的急剧增加也带来了巨大的挑战,尤其是在数据存储与处理的计算环境方面。
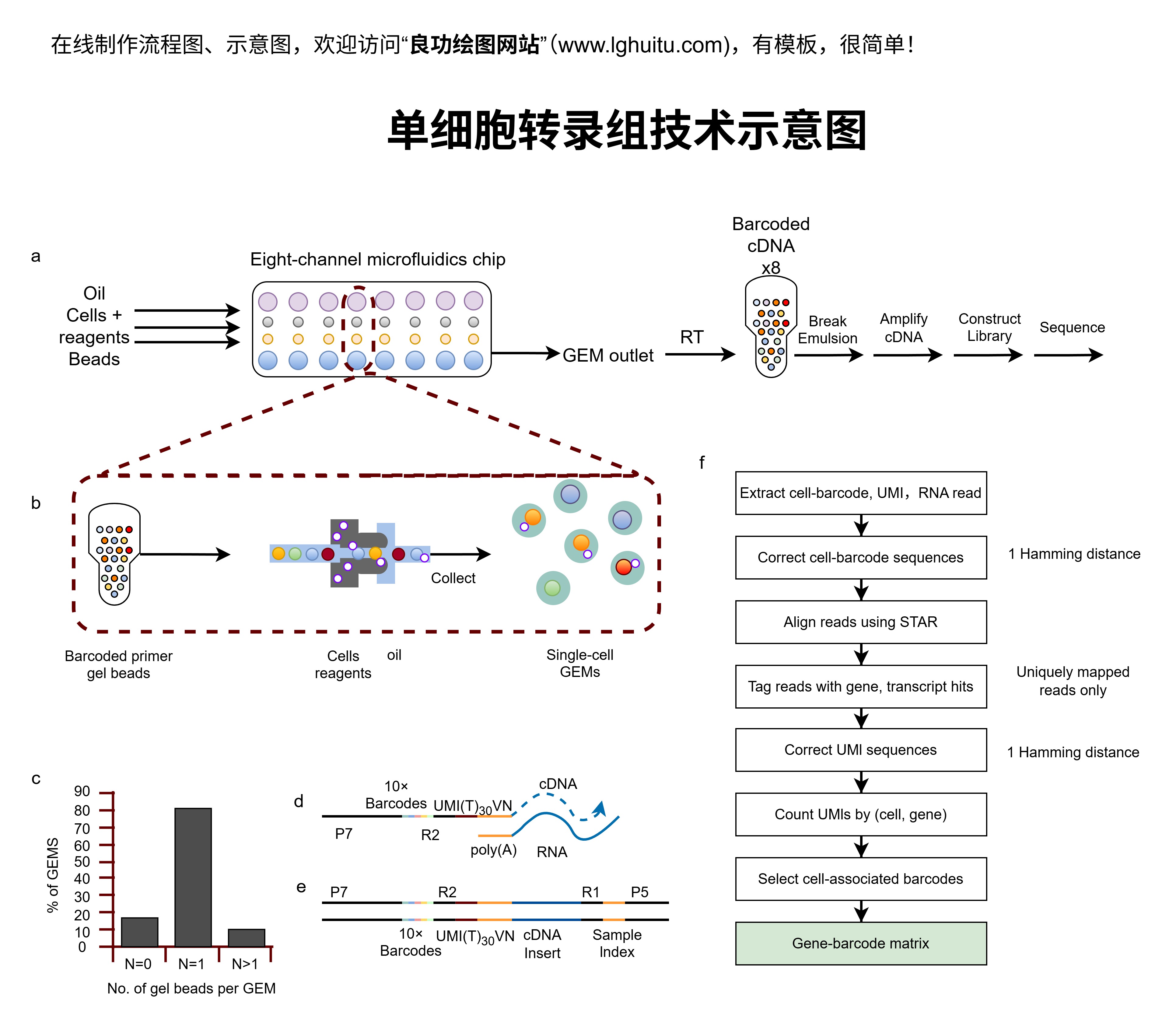
对于单细胞分析,研究人员面临的一个普遍问题就是“内存不够”。通常,单细胞分析的原始数据包含数百万个细胞,每个细胞的数据维度非常高。以单细胞RNA测序为例,原始数据的大小常常达到几十GB甚至更高。而这类数据的处理通常需要极高的计算资源,包括大内存和强大的处理器。尤其是在数据预处理、基因表达量计算、降维分析、聚类分析等阶段,计算资源的消耗极为显著。
面对内存不足的困境,研究人员如何应对呢?我们需要了解内存不足问题的根源。单细胞数据的稀疏性和高维度是主要原因之一。传统的数据分析方法往往不能高效处理这类大规模且高维度的数据,导致内存和计算资源的消耗异常巨大。因此,如何高效地利用现有计算资源,成为了研究人员迫切需要解决的问题。
一方面,很多传统的计算机硬件,如个人电脑和一般的工作站,并不具备处理大规模单细胞数据的能力。针对这一问题,研究人员通常选择在高性能计算集群(HPC)上运行分析任务,但即使如此,分析的过程也会因为内存不足而出现瓶颈。为了更好地应对这些挑战,越来越多的研究团队开始寻求优化计算资源的方案。
一种行之有效的方式是采用分布式计算架构。例如,通过将单细胞数据拆分成多个子任务并在不同的计算节点上并行处理,可以大大减轻单个节点的计算压力。这种方法不仅能节省内存,还能够提高数据处理的效率。分布式计算也对数据传输、任务调度等方面提出了更高的要求,需要研究人员在软硬件环境上进行充分的调配。
随着云计算技术的普及,云平台逐渐成为解决单细胞分析内存不足问题的一个重要选择。通过云服务提供商提供的高性能计算实例,研究人员可以根据需求灵活扩展计算资源,避免了硬件购置和维护的高额成本。例如,亚马逊AWS、谷歌云和微软Azure等云平台都为生物信息学研究提供了专门的计算资源和工具。云计算平台不仅能提供高内存、高性能的计算实例,还能根据项目需求进行弹性扩展,使研究人员能够在不增加硬件投入的情况下处理大规模数据。
除了硬件方面的优化,软件层面的改进同样不可忽视。在单细胞分析中,许多分析工具和算法都需要大量的内存和计算资源,因此开发更加高效的算法成为解决问题的关键。例如,一些新的数据处理工具采用了更加高效的内存管理策略,通过优化算法的内存使用,能够在有限的硬件条件下提高计算效率。
一个典型的例子是基于稀疏矩阵的计算方法。在单细胞RNA测序数据中,由于绝大多数基因在某些细胞中并未表达,数据的稀疏性非常明显。传统的数据存储方式会浪费大量的内存,而稀疏矩阵则通过仅存储非零元素,大大节省了内存和存储空间。许多单细胞分析工具,如Seurat和Scanpy,已经采用了这种稀疏矩阵的存储方式,大幅降低了内存需求。
数据压缩技术也是解决内存不足问题的一种有效手段。通过对单细胞数据进行压缩,可以减少数据占用的存储空间,降低计算机内存的负担。例如,采用高效的压缩算法,可以在保证数据准确性的前提下,压缩数据的存储空间,使得数据处理变得更加轻松。
除此之外,针对单细胞数据分析的算法也在不断进化。传统的聚类和降维算法往往存在计算复杂度高、内存消耗大的问题。近年来,许多研究者提出了更加高效的算法,通过降低计算复杂度,优化内存使用,使得单细胞数据分析变得更加高效。比如,t-SNE(t-distributedstochasticneighborembedding)降维方法虽然效果显著,但计算成本较高。新兴的UMAP(UniformManifoldApproximationandProjection)算法在降低内存消耗的保持了较高的降维效果,得到了越来越多研究人员的青睐。
解决单细胞分析中内存不足的问题,离不开软硬件的共同优化。无论是通过硬件升级、云计算平台的应用,还是通过算法优化和数据压缩技术,研究人员都在不断寻求更高效的解决方案。随着技术的不断进步,我们相信,未来单细胞分析的内存瓶颈问题将会得到有效缓解,推动生物学研究和医学诊疗的发展进入新的阶段。
正如每一项科技创新所面临的挑战,克服单细胞分析中的内存瓶颈,不仅是科研人员的责任,也是科技发展的必由之路。通过不断探索和创新,我们终将迎来更加高效、精准的单细胞分析时代。